Nový software pro vizualizaci molekul RNA

Britští vědci ve spolupráci s kolegy z Česka a USA představili novou metodu vizualizace sekundárních struktur RNA. Jejich systém využívá obsáhlou databázi vzorů, na jejichž základě dokáže predikovat a následně zobrazit 2D strukturu konkrétní molekuly.
Kyselina ribonukleová (RNA) představuje jednu z klíčových molekul v našich buňkách, kde zastává řadu důležitých funkcí. Molekuly RNA dokáží předávat genetické informace, kontrolovat a regulovat výrobu bílkovin a katalyzovat různé chemické reakce.
Na rozdíl od DNA a její známé dvoušroubovice, RNA se vyznačuje daleko variabilnějšími tvary. A právě od konkrétního tvaru molekul, které mohou být velké a složité, se často odvíjí funkce ribonukleové kyseliny. Schopnost tyto struktury efektivně předpovídat je proto pro badatele velmi důležitá.
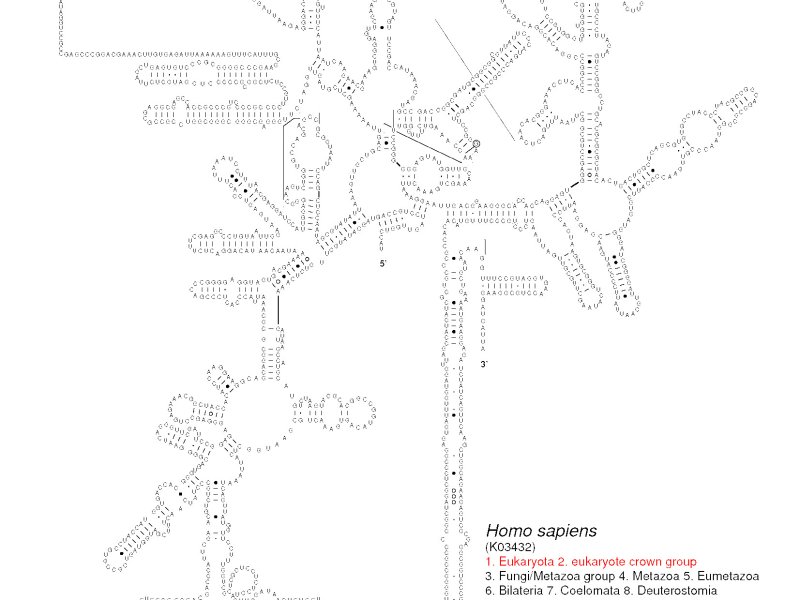
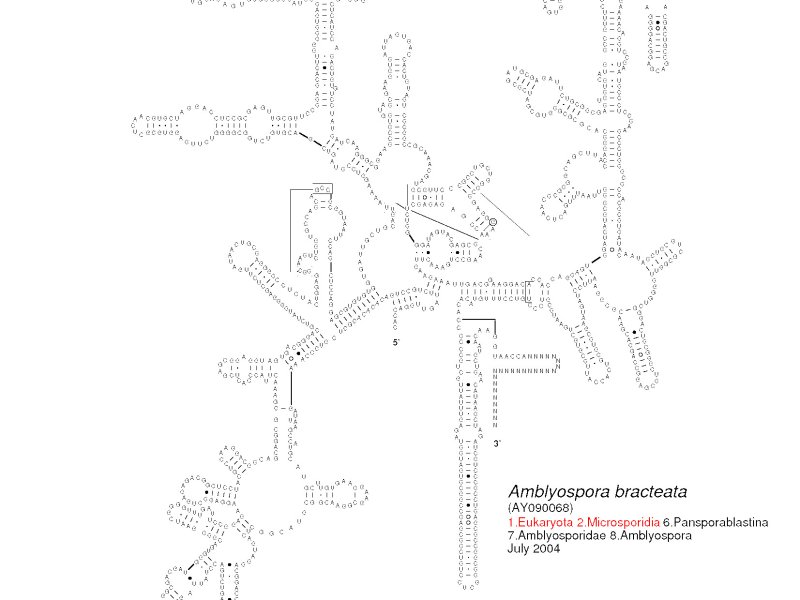
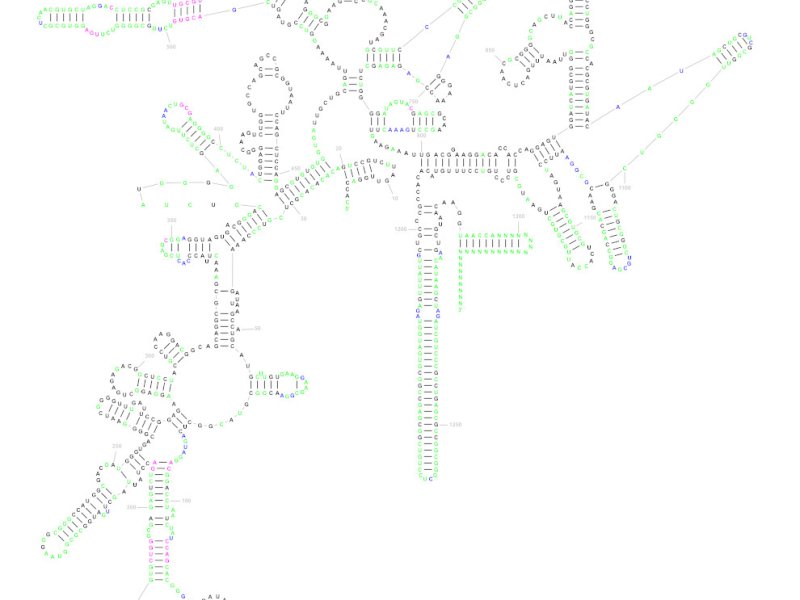
Mezinárodní tým složený z odborníků na informatiku a biologii stojí za vývojem softwaru R2DT, který na základě sekvence RNA dokáže predikovat sekundární strukturu dané molekuly a vizualizovat ji jako 2D diagram.
„Stejně jako v případě dalších makromolekul (DNA, proteiny) se molekula RNA skládá ze sekvence reziduí (nukleotidů), která v 3D prostoru zaujímá specifický, často evolučně konzervovaný tvar. Tento tvar je determinován intrareziduálními kontakty, které tvoří tzv. sekundární strukturu a tu lze vizualizovat jako 2D diagram,“ vysvětluje doc. David Hoksza z Katedry softwarového inženýrství MFF UK, spoluautor nové metody a související studie, kterou v červnu zveřejnil renomovaný časopis Nature Communications.
R2DT v sobě zahrnuje několik nástrojů včetně databáze, která obsahuje více než tři tisíce známých struktur RNA a jejich diagramů. Ty slouží jako šablony pro vygenerování sekundární struktury a diagramu pro vybranou RNA molekulu.
Vizualizace 2D je oproti 3D přehlednější a častěji prakticky využívána pro analýzu RNA molekul. Biologům se tak do rukou dostává nástroj poskytující zobrazení, na které jsou zvyklí a se kterým mohou jednoduše pracovat.
„Různé třídy RNA molekul, jako je ribozomální RNA nebo transferová RNA, mají standardní způsob, jak jsou vizualizovány, což umožňuje komparaci struktur z různých druhů, protože evolučně konzervované části molekul se ve vizualizaci nacházejí na stejném místě. Standardní diagramy bývají generovány (semi)automaticky, což znamená, že neexistuje algoritmus, který by říkal, jakým způsobem rozmístit nukleotidy tak, aby jejich pozice odpovídaly očekávání. Náš přístup pro vizualizaci funguje na principu existence vzoru, tj. molekuly, která je dostatečně evolučně blízká, aby se její sekundární struktura a její diagram dal použít pro vygenerování sekundární struktury a diagramu pro RNA molekulu, pro kterou tuto informaci nemáme,“ upřesňuje doc. Hoksza a dodává, že takový způsob vizualizace pomocí vzorů je zcela unikátní a žádný jiný software jej nevyužívá.
Člen mezioborové skupiny CUSBG (Charles University Structural Bioinformatics Group) a zástupce studijního programu Bioinformatika, společného programu Matfyzu a Přírodovědecké fakulty, doc. Hoksza, je autorem nástroje Traveler, který v rámci systému zajišťuje generování 2D diagramu. První verze tohoto nástroje vznikla ještě před startem projektu R2DT jako studentská bakalářská práce, kterou doc. Hoksza vedl.
Software R2DT, který vědci otestovali na vzorku čítajícím více než 13 milionů sekvencí RNA, je v současnosti plně funkční a volně dostupný prostřednictvím databáze RNAcentral a služby GitHub. Do budoucna chtějí vědci rozšířit databázi vzorů a přidat některá další vylepšení jako například možnost automatické kontroly kvality vizualizací.
OPMK, úvodní grafika Spencer Phillips/EMBL